Abteilung Siedlungswasserwirtschaft
Anwendung der abwasserbasierten Epidemiologie zum Nachweis von Atemwegsviren
Die Ausbreitung von Infektionskrankheiten wird traditionell mit klinischen Daten überwacht. Für viele Krankheitserreger ist die Erhebung klinischer Daten jedoch limitiert. Die abwasserbasierte Epidemiologie, welche den Fokus dieses Projekts darstellt, bietet einen ergänzenden Ansatz, um die Dynamik von Infektionskrankheiten unabhängig von der klinischen Überwachung zu verfolgen.
Wöchentliche Überwachung. Unser Ziel ist die Überwachung von viraler RNA wie SARS-Cov-2, Influenza A & B und Respiratory Syncytial Virus (RSV) im Abwasser verschiedener Kläranlagen in der ganzen Schweiz. Seit 2020 wurde ein 6-Plex-Assay mit der Technologie digital droplet -PCR (ddPCR) entwickelt, um die virale RNA für mehrere Targets zu quantifizieren. Die wöchentlich produzierten Daten werden auf dem WISE-Dashboard (Wastewater Surveillance) veröffentlicht. Die Erkenntnisse aus der abwasserbasierten Epidemiologie werden direkt mit dem Bundesamt für Gesundheit, das für Epidemien zuständig ist, und den relevanten Entscheidungsträgern geteilt (IDD - Infectious Diseases Dashboard). Damit liefern wir eine zusätzliche, datengestützte Grundlage für gesundheitspolitische Entscheidungen.
Inhalt und Ziele der Forschungsprojekte. Ziel dieses Projekts ist es, die Analysemethoden der abwasserbasierten Überwachung von Infektionskrankheiten, die wir während der Covid-19-Pandemie eingeführt haben, weiterzuentwickeln. Insbesondere erweitern wir unsere Arbeit auf andere Erreger (humane Coronaviren, Noroviren und andere enterische Krankheitserreger) und Methoden (z.B. Überwachung auf antimikrobiell resistente Bakterien durch Ausplattieren und Ganzgenomsequenzierung zum Nachweis von Signaturmutationen). Dabei wollen wir vor allem die folgenden drei Forschungsfragen beantworten:
- Welche Faktoren bestimmen die epidemiologische Ausbreitung von Viren? Wie früh können Virusausbrüche erkannt und die künftige Ausbreitung anhand von Abwasseranalysen modelliert werden?
- Können wir die genetischen Varianten, die eine epidemiologische Welle auslösen, schnell bestimmen? Können wir die zukünftige Entwicklung der Welle anhand von Abwasserdaten bestimmen?
- Wie verwandelt sich ein pandemisches Virus in ein endemisches Virus? Inwieweit können wir endemische Merkmale vorhersagen?
Historie
Projektverlauf
Update Januar 2025
Die die Reduktion der Anzahl überwachter Kläranlagen (ARA) immer noch die regionalen Trends der Virusbelastung im Abwasser ohne signifikanten Informationsverlust erfasst, reduzieren wir die Zahl der wöchentlich überwachten ARAs von 14 auf 10. Von nun an werden die Kläranlagen von Lugano, Zürich Werdhölzli, Lausanne Vidy, Chur, Sensetal Laupen, Basel, Genf Aire, Luzern Emmen/Buholz, Altenrhein und Neuchatel wöchentlich überwacht.
Update Juli 2023
Im Rahmen der Weiterführung des nationalen Abwasserüberwachungsprogramms wird die Eawag alle Analysen durchführen und von sechs auf 14 Kläranlagen aufstocken. In der Übergangszeit zwischen dem 29. Juni und dem 10. Juli werden aufgrund von logistischen Anpassungen und zusätzlicher Qualitätssicherung weniger oder keine Proben bearbeitet.
Update Dezember 2021
Wir messen im Abwasser den relativen Anteil (Prozentsatz) der Signaturmutationen der Delta-Variante (S:L452R) und der Alpha/Omicron-Varianten (HV69/70). Die Daten sind hier verfügbar und eine Visualisierung finden Sie bei den Dashboards.
Update Juni 2021
Auf Basis der Daten aus dem Abwasser schätzten unsere Projektpartner an der ETHZ (Gruppe cEvo, BSSE) die effektive Reproduktionszahl Re, d.h. die Anzahl an Personen eine infizierte Person im Laufe der Zeit ansteckt. Eine Visualisierung dieser Abschätzungen werden hier im direkten Vergleich zu den Abschätzungen basierend auf klinischen Fallzahlen gezeigt.
Update Mai 2021
Extrakte der Abwasserproben werden am FGCZ sequenziert. Mit Bioinformatik Tools schätzen unsere ETHZ Projektpartner (Gruppe Computational Biology, BSSE) das Vorkommen verschiedener Mutationen von SARS-CoV-2. Die Resultate von sechs Kläranlagen für B.1.1.7, B.1.351, P.1 and B.1.617* sind hier zusammengestellt.
Update April 2021
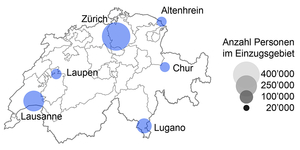
Im Februar 2021 startete das Projekt AbwasSARS-CoV-2 in Zusammenarbeit mit dem BAG (siehe Point de Presse vom 9. März 2021, Link zu Video ab Minute 15:36). In diesem Projekt wird täglich Rohabwasser von sechs Kläranlagen (siehe Abbildung 2) gesammelt und auf RNA von SARS-CoV-2 analysiert (Resultate).
Abbildung 2: Standorte der sechs Kläranlagen mit Angabe zur Grösse der Einzugsgebiete die im BAG-Projekt AbwasSARS-CoV-2 untersucht werden. Insgesamt sammeln und reinigen diese Kläranlagen zusammen das Abwasser von knapp 1 Million Einwohner.
Update Oktober 2020
Nach einer intensiven Zeit mit Methodenentwicklung und Datensammlung können wir jetzt folgendes zur Verfügung stellen:
Beinahe Echtzeit Messungen von den zwei Kläranlagen ARA Werdhölzli in Zürich und STEP Vidy in Lausanne: Grafik für Zürich und Grafik für Lausanne [nur Englisch; diese Grafiken werden nach dem Update im April nicht mehr aktualisiert, siehe neue Links oben]
Unsere Protokolle für Probenvorbereitung: ultrafiltration protocol (neue Version hier) und extraction protocol [nur Englisch]
Antworten auf häufig gestellte Fragen (siehe Link zu FAQ)
Ursprüngliche Projektbeschreibung Juni 2020
Anwendung der abwasserbasierten Epidemiologie zur Erkennung von SARS-CoV-2
Abwasser bietet potenzielle Erkenntnisse in ein besseres Verständnis der Krankheitslast und der Krankheitsdynamik der aktuellen COVID-19-Pandemie. Obwohl es sich bei COVID-19 um eine Atemwegserkrankung handelt, scheidet ein erheblicher Teil der Menschen Erbgut des Virus (RNA) im Stuhl aus. Durch das Sammeln und Analysieren von Abwasserproben auf dieses Erbgut hin können wir feststellen, ob und in welchem Ausmass Menschen im Einzugsgebiet einer Kläranlage mit dem Virus infiziert sind. Aufgrund der zeitlichen Verzögerung zwischen Symptomen und bestätigten Testergebnissen können wir möglicherweise das Virus, das über das Abwasser in den Gemeinden zirkuliert, nachweisen, bevor klinische Falldaten verfügbar sind.
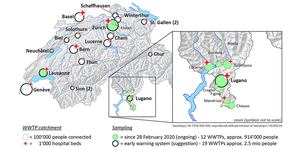
Abbildung 1: Am Anfang der ersten Welle, sammelten wir Proben von 12 Kläranlagen (grüne Kreise) und machten mit Lausanne und Zürich weiter. Proben von 19 grossen Kläranlagen (dick umrandete Kreise) würden 2.5 Millionen Personen umfassen.
In dieser Studie untersuchen wir (die Labors von Tim Julian und Christoph Ort, Eawag; Tamar Kohn und Xavier Fernandez Cassi, EPFL) Abwasserproben aus Lausanne, Zürich und dem Tessin auf das Vorhandensein und die Menge von SARS-CoV-2 vom Beginn des Ausbruchs (Ende Februar) bis heute. Wir werden die Daten in Modelle des Krankheitsverlaufs integrieren, in der Hoffnung, dass wir zu Erkenntnissen über die Krankheit in der Schweiz beitragen können. Darüber hinaus werden wir Methoden für den Nachweis von SARS-CoV-2 bei sehr niedrigen Konzentrationen entwickeln und verbessern, um zur Entwicklung und Verbesserung eines Frühwarnsystems für das Auftreten oder Wiederauftreten der Krankheit beizutragen.
Archivierte Dashboards
Wissenschaftliche Berichte
Aktuelle Reports sind auf der Website des WISE-Projekts in der Rubrik «Reports» zu finden: Wastewater Surveillance
Videos & Printmedien
Aktuelle Beiträge aus Printmedien und Videos sind auf der Website des WISE-Projekts unter der Rubrik «Printmedia» und «Videos» zu finden: Wastewater Surveillance