Abteilung Siedlungswasserwirtschaft
SARS-CoV-2 im Abwasser
News
4. Juli 2023: Im Rahmen der Weiterführung des nationalen Abwasserüberwachungsprogramms wird die Eawag alle Analysen durchführen und von sechs auf 14 Kläranlagen aufstocken. In der Übergangszeit zwischen dem 29. Juni und dem 10. Juli werden aufgrund von logistischen Anpassungen und zusätzlicher Qualitätssicherung weniger oder keine Proben analysiert.
Startet im November 2022: Wastewater-based Infectious Disease Surveillance WISE SNSF Sinergia Projekt (in Zusammenarbeit mit ETHZ und EPFL)
Die Ausbreitung von Infektionskrankheiten wird traditionell mit klinischen Daten überwacht. Bei vielen Krankheitserregern ist die klinische Datenerfassung jedoch unzureichend. Die abwasserbasierte Epidemiologie, auf welche in diesem Projekt fokussiert wird, bietet einen ergänzenden Ansatz, um die Dynamik von Infektionskrankheiten unabhängig von der klinischen Überwachung zu verfolgen.
Inhalt und Ziele des Forschungsprojekts. Ziel dieses Projekts ist es, die Methoden und Analysen der abwasserbasierten Überwachung von Infektionskrankheiten, welche wir während der Covid-19 Pandemie etabliert haben, weiterzuentwickeln. Insbesondere werden wir unsere Arbeit auf weitere Krankheitserreger (humane Coronaviren, Influenza, Noroviren und andere Enteropathogene) ausdehnen. Damit wollen wir primär folgende drei Forschungsfragen beantworten:
- Welche Faktoren bestimmten die epidemiologische Ausbreitung von Viren? Wie früh können Virusausbrüche erkannt und die zukünftige Ausbreitung anhand von Abwasseranalysen modelliert werden?
- Können wir die genetischen Varianten, die eine epidemiologische Welle starten, schnell bestimmen? Können wir die künftige Entwicklung der Welle auf der Grundlage von Abwasserdaten bestimmen?
- Wie verwandelt sich ein pandemisches Virus in ein endemisches Virus? Inwieweit können wir endemische Eigenschaften vorhersagen?
Wissenschaftlicher und gesellschaftlicher Kontext des Forschungsprojekts. Wir werden Erkenntnisse aus der abwasserbasierten Epidemiologie direkt an das für Epidemien zuständige Bundesamt für Gesundheit und die relevanten Entscheidungsträger weitergeben. Somit bieten wir für die hier überwachten Aspekte eine weitere daten-gestützte Grundlage für gesundheitspolitische Entscheidungen. Generell werden wir unterstützen falls die Forschung zur abwasserbasierten Epidemiologie in ein schweizweites Überwachungssystem überführt werden soll.
Projektbeschreibung und Verlauf
Projektbeschreibung
Abwasserbasierte Epidemiologie gewährt unabhängige Einblicke in die Virenlast und Dynamik der gegenwärtigen COVID-19 Pandemie. Durch die Überwachung von SARS-CoV-2 RNA im Zulauf (oder Rohabwasser) von Schweizer Kläranlagen komplementieren Abwasserdaten epidemiologische Indikatoren wie Fallzahlen und ergeben so ein umfassenderes Bild des Pandemieverlaufs. Die Analyse von SARS-CoV-2 RNA im Abwasser ermöglicht ausserdem einen Überblick über die Zirkulation von problematischen Varianten durch gezielten Nachweis von Signaturmutationen sowie durch Whole-Genome-Sequencing. In diesem Projekt werden Methoden für den empfindlichen und spezifischen Nachweis von SARS-CoV-2 und anderen Krankheitserregern im Abwasser entwickelt, angewandt, bewertet und weiterentwickelt, um zusätzliche Informationen über Krankheitsdynamiken zu erhalten.
Projektverlauf
Update Dezember 2021
Wir messen im Abwasser den relativen Anteil (Prozentsatz) der Signaturmutationen der Delta-Variante (S:L452R) und der Alpha/Omicron-Varianten (HV69/70). Die Daten sind hier verfügbar und eine Visualisierung finden Sie bei den Dashboards.
Update Juni 2021
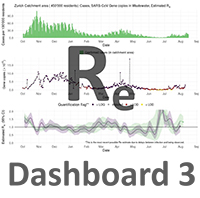
Auf Basis der Daten aus dem Abwasser schätzten unsere Projektpartner an der ETHZ (Gruppe cEvo, BSSE) die effektive Reproduktionszahl Re, d.h. die Anzahl an Personen eine infizierte Person im Laufe der Zeit ansteckt. Eine Visualisierung dieser Abschätzungen werden hier im direkten Vergleich zu den Abschätzungen basierend auf klinischen Fallzahlen gezeigt.
Update Mai 2021
Extrakte der Abwasserproben werden am FGCZ sequenziert. Mit Bioinformatik Tools schätzen unsere ETHZ Projektpartner (Gruppe Computational Biology, BSSE) das Vorkommen verschiedener Mutationen von SARS-CoV-2. Die Resultate von sechs Kläranlagen für B.1.1.7, B.1.351, P.1 and B.1.617* sind hier zusammengestellt.
Update April 2021
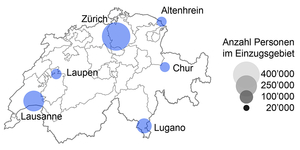
Im Februar 2021 startete das Projekt AbwasSARS-CoV-2 in Zusammenarbeit mit dem BAG (siehe Point de Presse vom 9. März 2021, Link zu Video ab Minute 15:36). In diesem Projekt wird täglich Rohabwasser von sechs Kläranlagen (siehe Abbildung 2) gesammelt und auf RNA von SARS-CoV-2 analysiert (Resultate).
Abbildung 2: Standorte der sechs Kläranlagen mit Angabe zur Grösse der Einzugsgebiete die im BAG-Projekt AbwasSARS-CoV-2 untersucht werden. Insgesamt sammeln und reinigen diese Kläranlagen zusammen das Abwasser von knapp 1 Million Einwohner.
Momentan werden die Messreihen in der Regel ein- bis zweimal pro Woche aktualisiert (abhängig von Transport und Verarbeitungskapazität erfolgt das Update nicht für alle Standorte am gleichen Tag). Mit folgenden Links gelangen Sie zu den Grafiken:
Übersicht alle Kläranlagen
Altenrhein ARA Altenrhein
Chur ARA Chur
Laupen ARA Sensetal
Lausanne STEP Vidy
Lugano CDA Lugano
Zürich ARA Werdhölzli
Update Oktober 2020
Nach einer intensiven Zeit mit Methodenentwicklung und Datensammlung können wir jetzt folgendes zur Verfügung stellen:
Beinahe Echtzeit Messungen von den zwei Kläranlagen ARA Werdhölzli in Zürich und STEP Vidy in Lausanne: Grafik für Zürich und Grafik für Lausanne [nur Englisch; diese Grafiken werden nach dem Update im April nicht mehr aktualisiert, siehe neue Links oben]
Unsere Protokolle für Probenvorbereitung: ultrafiltration protocol (neue Version hier) und extraction protocol [nur Englisch]
Antworten auf häufig gestellte Fragen (siehe Link zu FAQ)
Ursprüngliche Projektbeschreibung Juni 2020
Anwendung der abwasserbasierten Epidemiologie zur Erkennung von SARS-CoV-2
Abwasser bietet potenzielle Erkenntnisse in ein besseres Verständnis der Krankheitslast und der Krankheitsdynamik der aktuellen COVID-19-Pandemie. Obwohl es sich bei COVID-19 um eine Atemwegserkrankung handelt, scheidet ein erheblicher Teil der Menschen Erbgut des Virus (RNA) im Stuhl aus. Durch das Sammeln und Analysieren von Abwasserproben auf dieses Erbgut hin können wir feststellen, ob und in welchem Ausmass Menschen im Einzugsgebiet einer Kläranlage mit dem Virus infiziert sind. Aufgrund der zeitlichen Verzögerung zwischen Symptomen und bestätigten Testergebnissen können wir möglicherweise das Virus, das über das Abwasser in den Gemeinden zirkuliert, nachweisen, bevor klinische Falldaten verfügbar sind.
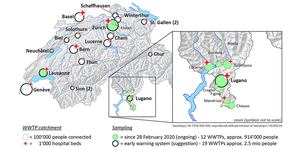
Abbildung 1: Am Anfang der ersten Welle, sammelten wir Proben von 12 Kläranlagen (grüne Kreise) und machten mit Lausanne und Zürich weiter. Proben von 19 grossen Kläranlagen (dick umrandete Kreise) würden 2.5 Millionen Personen umfassen.
In dieser Studie untersuchen wir (die Labors von Tim Julian und Christoph Ort, Eawag; Tamar Kohn und Xavier Fernandez Cassi, EPFL) Abwasserproben aus Lausanne, Zürich und dem Tessin auf das Vorhandensein und die Menge von SARS-CoV-2 vom Beginn des Ausbruchs (Ende Februar) bis heute. Wir werden die Daten in Modelle des Krankheitsverlaufs integrieren, in der Hoffnung, dass wir zu Erkenntnissen über die Krankheit in der Schweiz beitragen können. Darüber hinaus werden wir Methoden für den Nachweis von SARS-CoV-2 bei sehr niedrigen Konzentrationen entwickeln und verbessern, um zur Entwicklung und Verbesserung eines Frühwarnsystems für das Auftreten oder Wiederauftreten der Krankheit beizutragen.
Nationales Abwassermonitoring BAG
Die Daten des nationalen Abwassermonitorings des BAG werden seit Juli 2022 auf dem BAG Dashboard Viruslast im Abwasser publiziert.
Publikationen
Cariti, F., Tuñas Corzon, A., Fernandez-Cassi, X., Ganesanandamoorthy, P., Ort, C., Julian, T. R., & Kohn, T. (2022). Wastewater reveals the spatiotemporal spread of SARS-CoV-2 in the Canton of Ticino (Switzerland) during the onset of the COVID-19 pandemic. ACS ES&T Water. https://doi.org/10.1021/acsestwater.2c00082
Jahn, K., Dreifuss, D., Topolsky, I., Kull, A., Ganesanandamoorthy, P., Fernandez-Cassi, X., … Beerenwinkel, N. (2022). Early detection and surveillance of SARS-CoV-2 genomic variants in wastewater using COJAC. Nature Microbiology, 7, 1151-1160. https://doi.org/10.1038/s41564-022-01185-x
Huisman, J.S., Scire, J., Caduff, L., Fernandez-Cassi, X., Ganesanandamoorthy, P., Kull, A., Scheidegger, A., Stachler, E., Boehm, A.B., Hughes, B., Knudson, A., Topol, A., Wigginton, K.R., Wolfe, M.K., Kohn, T., Ort, C., Stadler, T., Julian, T.R. (2022). Wastewater-Based Estimation of the Effective Reproductive Number of SARS-CoV-2. Environ. Health Perspect. 130:5. https://doi.org/10.1289/EHP10050
Caduff L., Dreifuss D., Schindler T., Devaux A.J., Ganesanandamoorthy P., Kull A., Stachler E., Fernandez-Cassi X., Beerenwinkel N., Kohn T., Ort C., Julian T.R. (2022). Inferring transmission fitness advantage of SARS-CoV-2 variants of concern from wastewater samples using digital PCR, Switzerland, December 2020 through March 2021. Euro Surveill. 27(10). https://doi.org/10.2807/1560-7917.ES.2022.27.10.2100806
Fernandez-Cassi X., Scheidegger A., Bänziger C., Cariti F., Tuñas Corzon A., Ganesanandamoorthy P., Lemaitre J.C., Ort C., Julian T.R., Kohn T. (2021). Wastewater monitoring outperforms case numbers as a tool to track COVID-19 incidence dynamics when test positivity rates are high. Water Res. 200: 117252. https://doi.org/10.1016/j.watres.2021.117252
Archivierte Dashboards
Team












Wissenschaftlicher Bericht 2022-2023
Medien
Videos
SNF «Covid-19, NFP 78» (12. Juli 2023)
"Nano" auf 3Sat (4. Februar 2022)
SRF “Kinder-News” (22. Februar 2021)
SRF “Einstein” (30. April 2020)
französisch
"RTS" Des tests sont effectués dans les eaux usées pour déterminer la présence ou non du coronavirus dans la population (20. April 2022)
RTS "Le coronavirus est détectable dans les eaux usées, selon une étude" (30. Avril 2020)
Printmedien
Tagesanzeiter "Schweiz diskutiert Testpflicht für Einreisende aus China" (10. Januar 2023, Abo)
Tagesanzeiger "Welche Corona-Zahlen jetzt noch wichtig sind" (6. April 2022, Abo)
Tagesanzeiger "BAG lässt Corona-Forscher hängen" (31. März 2022, Abo)
Blick “Traum von Herdenimmunität geplatzt” (19. März 2022)
Tagesanzeiger “Coronavirus in Zürich“ (7. Januar 2022, Abo)
Blick “Delta kann sich im Abwasser nicht verstecken” (29. Juni 2021)
Tagesanzeiger “Coronavirus im Abwasser” (30. April 2021)
Blick “Virenjäger in der Kläranlage” (10. März 2021)
französisch
Ville de Lausanne "Surveillance du coronavirus dans les eaux usées"
Swissinfo "Ils traquent la Covid-19 dans les eaux usées" (14. Mai 2021)
24 heures "Lausanne surveille la pandémie grâce aux eaux usées" (17. Décembre 2020)